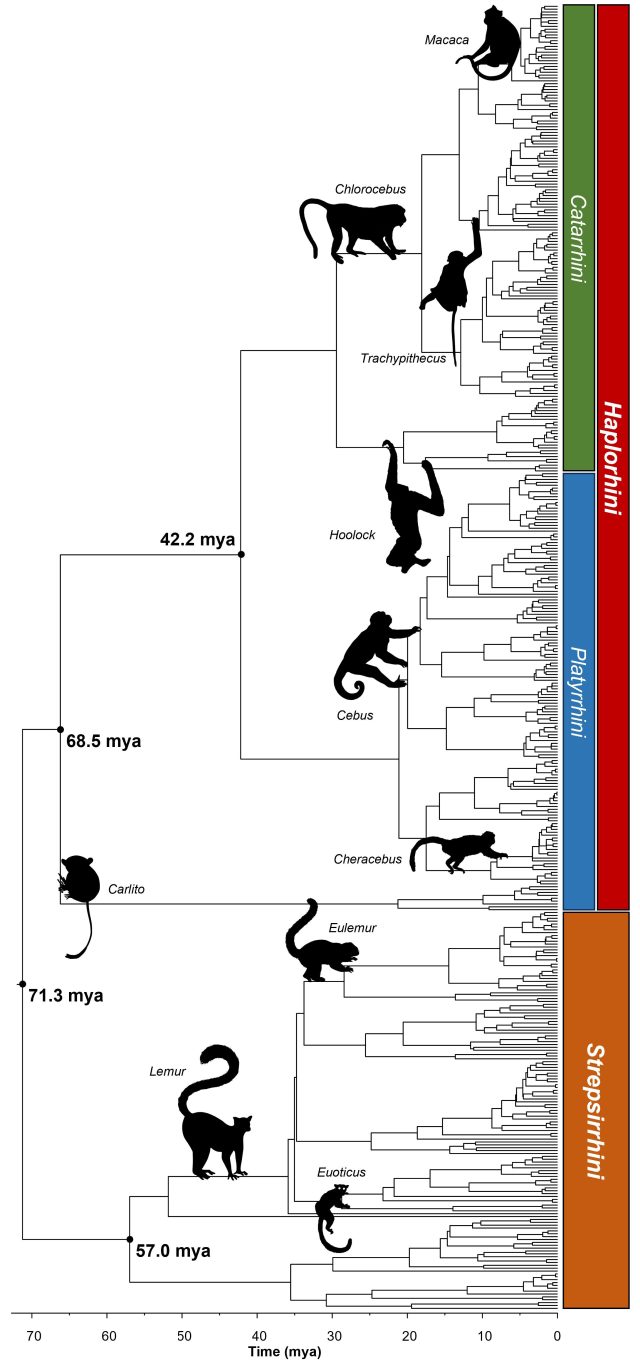
Drzewo czasu 455 gatunków naczelnych. Zdjęcia z phylopic.org. Źródło: Craig i in., 2024.
W nowym artykule opublikowany W Granice w bioinformatycebiolodzy dr Jack M. Craig, dr Blair Hedges i dr Sudhir Kumar z Temple University zbudowali drzewo ewolucyjne obejmujące 455 naczelnych, każdy gatunek, dla którego dostępne są dane genetyczne. Drzewo, najbardziej kompletne w swoim rodzaju, przedstawia ewolucyjną skalę czasową całego rzędu naczelnych, w tym małp, małp człekokształtnych, lemurów, lorysów i galagów.
W poniższym gościnnym artykule redakcyjnym dr Craig opisuje etapy uzyskiwania niemal kompletnego drzewa czasu dla naczelnych i wyjaśnia wartość takich danych:
Zakon naczelnych składa się nie tylko z naszych najbliższych krewnych na Ziemi, siedmiu wielkich małp człekokształtnych, ale także z ponad 450 gatunków małp, lemurów, lorysów i galagów. Naczelne są fantastycznie różnorodne, od goryli ważących 400 funtów po lemury mysie (Microcebus) ważące zaledwie jedną uncję. Wykazują jedne z najbardziej niezwykłych zachowań obserwowanych w przyrodzie; szympansy „łowieją” termity w wydrążonych kłodach, używając specjalnie dobranych patyków, podczas gdy orangutany używają liści jako rękawiczek do obchodzenia się z kolczastymi owocami duriana.
Są to jedne z najintensywniej badanych gatunków na Ziemi, a mimo to nie istnieje kompleksowa hipoteza filogenetyczna molekularna dotycząca historii ewolucji naczelnych, która podsumowywałaby wzór i czas trwania wszystkich pokrewieństw między naczelnymi.
Takie drzewo filogenetyczne wykorzystywałoby dane dotyczące sekwencji molekularnej, aby powiedzieć nam zarówno, kiedy każdy gatunek lub grupa gatunków pojawiła się po raz pierwszy, jak i które inne grupy na drzewie są ich najbliższymi krewnymi. Największe czasowe molekularne drzewo filogenetyczne życia, zwane „drzewom czasu”, obejmuje nieco ponad 200 gatunków naczelnych, podczas gdy największe syntetyczne drzewo czasu, oparte na ponad 4000 opublikowanych badań, obejmuje zaledwie dwukrotnie tę liczbę, pozostawiając około jednej piątej naczelnych drzewo życia nierozwiązane.
Dlaczego potrzebujemy kompletnych drzew ewolucyjnych
Nie można niedocenić wartości drzew ewolucyjnych zsynchronizowanych w czasie, zawierających każdy gatunek z danej linii. Chociaż takie drzewa są z natury fascynujące, ponieważ ukazują ewolucyjną historię, która dała nam obecną różnorodność biologiczną, tworzą one również istotne podstawy dla wielu rodzajów przyszłych prac.
Na przykład taksonomiczne i systematyczne wysiłki mające na celu katalogowanie gatunków polegają na tym, że identyfikują nowe linie rodowe. Badania tempa ewolucji i jej możliwych korelatów, takich jak zmiany klimatyczne i geologiczne, są zasadniczo powiązane z leżącymi u ich podstaw filogenezami.
Dziedziny takie jak biogeografia, filogeografia i ekologia historyczna, które wykorzystują drzewa czasu do badania wzorców przestrzennych lub ekologicznych, byłyby niemożliwe bez filogenezy. A gdy obserwujemy zanikanie globalnej różnorodności biologicznej w wyniku trwającego wymierania, filogenezy stają się niezbędnymi narzędziami w identyfikowaniu priorytetów ochrony i ocenie skutków naszych wysiłków na rzecz ochrony gatunków.
Jak powszechne są filogenezy pełne?
Ponieważ kompleksowe filogenezy molekularne są cennymi narzędziami, zaskakującym może być fakt, że są one rzadkie. Baza danych taksonomii NCBI zawiera obecnie sekwencje molekularne prawie 500 000 gatunków, natomiast Drzewo Czasu Życianajwiększa baza danych opublikowanych filogenez czasowych, obejmuje około 150 000 gatunków.
Przeglądając zbiór badań zawartych w bazie danych, odkryliśmy, że większość filogenezy jest zwykle niewielka i obejmuje średnio tylko 25 gatunków. Drzewa te są dziełem ludzi zajmujących się badaniem grup blisko spokrewnionych organizmów, takich jak rodzaje lub rodziny, dla których w ich systemie badań priorytetem jest rozwiązanie na szerszą skalę. Zatem potrzeba kompletnego drzewa życia zostanie zaspokojona tylko wtedy, gdy znajdziemy sposób na połączenie tych wysiłków.
Odkryj najnowsze osiągnięcia nauki, technologii i kosmosu dzięki over 100 000 abonentów którzy codziennie korzystają z witryny Phys.org. Zapisz się do naszego bezpłatny biuletyn i otrzymuj aktualne informacje o przełomowych, innowacjach i badaniach, które mają znaczenie—codziennie lub co tydzień.
Nowa droga naprzód
Chociaż duże, w pełni zsynchronizowane drzewa posiadające dane dotyczące sekwencji molekularnej wszystkich gatunków są rzadkie, odkryliśmy, że materiały do ich budowy są powszechne. Po pierwsze, filogenezy nieograniczone w czasie znacznie przewyższają liczbę filogenezy czasowych w literaturze, nawet wśród artykułów opublikowanych w ciągu ostatnich dziesięciu lat. Wystarczy jedna lub kilka kalibracji, aby stać się cennymi składnikami globalnego drzewa czasu życia.
Chociaż wiele gatunków nigdy nie zostało włączonych do filogenezy molekularnej, często odpowiednie dane molekularne są zdeponowane w repozytoriach takich jak NCBI GenBank, gdzie badacze mają swobodny dostęp do informacji o sekwencji DNA. Te dwa źródła danych stanowią fantastyczną okazję do zbudowania kompleksowych drzew czasu.
W ciągu kilku publikacji opracowaliśmy podejście do budowania superdrzewa, obejmujące połączenie wszystkich opublikowanych filogenez w określonym czasie, w tym interesujących gatunków; poszukiwanie drzew nieokreślonych w czasie, w tym wszelkich pozostałych gatunków, a następnie nowatorskie określenie czasu przy użyciu wtórnych kalibracji zgodnych z literaturą; i wreszcie zestawienie dopasowań de novo i ostatecznie filogenezy w czasie w oparciu o publicznie dostępne dane.
W ramach naszych ostatnich wysiłków poszukiwania te odsłoniły wystarczającą ilość danych, aby zbudować nowe syntetyczne superdrzewo składające się z 455 naczelnych, czyli 98% wszystkich występujących w taksonomii NCBI i o 55 więcej niż było już obecnych w TimeTree. Nasze nowe drzewo czasu stanowi jak dotąd najpełniejszy opis powiązań ewolucyjnych między naczelnymi.
Ukończenie Drzewa Czasu Życia
Wysiłki te pokazały, że chociaż historia ewolucji nawet niektórych z najbardziej charyzmatycznych gatunków na Ziemi pozostaje nie do końca poznana, dysponujemy narzędziami pozwalającymi wypełnić znaczną część tej luki w wiedzy. Wyobrażamy sobie nasz protokół badawczy jako dostępne i ostatecznie niezwykle cenne narzędzie w naszych wysiłkach na rzecz zrozumienia ewolucji. Kompletne drzewa czasu są podstawowym zasobem w wielu dziedzinach i odkryliśmy, że często można je zbudować na podstawie istniejących danych.
Co więcej, takie kompletne drzewa czasu pozwalają nam testować hipotezy, których inaczej nie moglibyśmy przetestować. Na przykład w niniejszym badaniu sprawdziliśmy, czy liczbę gatunków w różnych kladach naczelnych można lepiej wyjaśnić unikalnym tempem specjacji, przy czym niektóre linie naczelnych generują nowe gatunki znacznie szybciej niż inne, czy też najlepszym wyjaśnieniem jest po prostu czas, wszystkie linie tworzą nowe gatunki w mniej więcej tym samym tempie, a starsze linie tworzą z czasem więcej gatunków.
Odkryliśmy, że w rzeczywistości wszystkie główne grupy naczelnych charakteryzowały się stosunkowo podobnym tempem specjacji, w związku z czym ich wiek był lepszym prognostykiem bogactwa gatunkowego. Analiza ta byłaby dość problematyczna, gdybyśmy w naszym drzewie czasu brakowało wielu gatunków lub dat, dlatego służy jako doskonały przykład użyteczności dużych, kompletnych drzew czasu.
Więcej informacji:
Uzupełnianie molekularnego drzewa czasu naczelnych, Granice w bioinformatyce (2024). DOI: 10.3389/fbinf.2024.1495417. frontiersin.org/news/2024/12/1 … nary-history-of-life
Cytat: Uzupełnianie „drzewa czasu” naczelnych: nowy sposób mapowania ewolucyjnej historii życia na Ziemi (2024, 16 grudnia) pobrano 16 grudnia 2024 r. z https://phys.org/news/2024-12-timetree-primates- historia-ewolucyjna-życie.html
Niniejszy dokument podlega prawom autorskim. Z wyjątkiem uczciwego obrotu w celach prywatnych studiów lub badań, żadna część nie może być powielana bez pisemnej zgody. Treść jest udostępniana wyłącznie w celach informacyjnych.