PELSA to czuła, wszechstronna metoda identyfikacji białek i miejsc wiążących ligandy w układach natywnych bez konieczności modyfikacji ligandów. Przewyższa tradycyjne podejścia pod względem czułości, szczególnie w przypadku interakcji metabolitów o niskim powinowactwie i oferuje szerokie zastosowania w projektowaniu leków i badaniach biologicznych.
Zespół badawczy kierowany przez prof. Minglianga Ye z Dalian Institute of Chemical Physics Chińskiej Akademii Nauk (CAS), we współpracy z grupą prof. Cheng Luo z Shanghai Institute of Materia Medica, CAS, opracował bardzo czułą technikę proteomiczną znany jako test stabilności lokalnej skupiony na peptydach (PELSA). Opublikowano w Metody naturyta innowacyjna metoda umożliwia jednoczesną identyfikację białek wiążących ligandy i miejsc ich wiązania w złożonych układach. Metoda PELSA jest bardzo wszechstronna i ma zastosowanie do szerokiej gamy ligandów, takich jak metabolity, leki i zanieczyszczenia.
Interakcje białek z ligandami – działającymi jako substraty enzymów, inhibitory, cząsteczki sygnalizacyjne, modulatory allosteryczne lub kotwice strukturalne – mają fundamentalne znaczenie dla ich funkcji biochemicznych.
Charakterystyka interakcji białko-ligand ma kluczowe znaczenie dla identyfikacji funkcji niescharakteryzowanych białek, badania mechanizmów regulacyjnych w metabolizmie komórkowym i zrozumienia mechanizmów działania leków. Ponadto określenie regionów wiążących ligand jest nieocenione przy projektowaniu leków w oparciu o strukturę i formułowaniu hipotez biologicznych.
Ograniczenia metod tradycyjnych
Tradycyjne metody określania miejsc wiązania i powinowactwa zazwyczaj wymagają oczyszczania rekombinowanych białek, co może być zarówno czasochłonne, jak i pracochłonne. Ponadto oczyszczone białka mogą nie w pełni replikować swój natywny stan komórkowy, co skutkuje niedokładnymi pomiarami powinowactwa. Metody proteomiki oparte na modyfikacjach stanowią skuteczne rozwiązanie do identyfikacji białek wiążących ligandy i ich miejsc bezpośrednio w natywnych lizatach komórkowych. Jednakże często wymagają modyfikacji ligandów, co może wpływać na aktywność ligandów i nie można ich zastosować do ligandów, których nie można modyfikować.
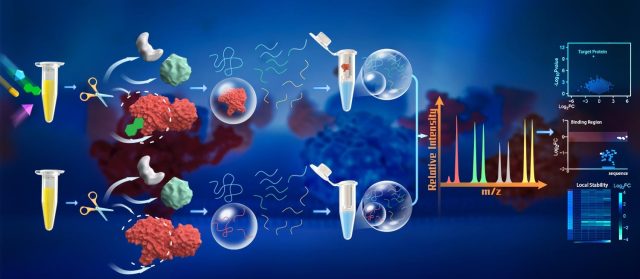
W metodzie zaproponowanej w tym badaniu badacze wykorzystali dużą ilość trypsyny (stosunek E/S wynoszący 1:2) do bezpośredniego wytworzenia małych peptydów z natywnych białek. Ponieważ peptydy te powstają w warunkach natywnych, ich liczebność stanowiła miarę lokalnej stabilności białek.
Duża ilość trypsyny umożliwiła rozszczepienie nawet segmentów białek o niskim stanie energetycznym, co doprowadziło do wygenerowania dużej liczby peptydów odzwierciedlających lokalną stabilność białka. Peptydy te oddzielono od częściowo strawionych białek, zebrano i bezpośrednio analizowano metodą spektrometrii mas. Mierząc liczebność peptydu w próbkach traktowanych ligandem i nośnikiem, można następnie określić regiony wiążące ligand i odpowiadające im białka wiążące.
Zwiększona czułość i niezawodność
Test PELSA wykazał wyższą czułość w identyfikacji białek docelowych. Na przykład, identyfikując białka docelowe staurosporyny, inhibitora pankinazy, badanie PELSA wykazało 12-krotny wzrost identyfikacji docelowych kinaz w porównaniu z najnowocześniejszą metodą bez modyfikacji, LiP-MS. W porównaniu z szeroko stosowaną techniką termicznego profilowania proteomów (TPP), w której brakuje informacji o miejscu wiązania, w ramach projektu PELSA zidentyfikowano 2,4-krotnie więcej celów kinazowych. Zależne od dawki eksperymenty PELSA mogą mierzyć lokalne powinowactwo, dostarczając wglądu w dynamiczne zmiany strukturalne białek po związaniu ligandu w warunkach fizjologicznych.
Metabolity, znane ze swojej różnorodności strukturalnej i często niskiego powinowactwa wiązania z białkami, stwarzają wyzwania. Metoda PELSA okazała się szczególnie skuteczna w systematycznej identyfikacji białek wiążących metabolity. Na przykład w ramach projektu PELSA zidentyfikowano 40 potencjalnych białek docelowych dla alfa-ketoglutaranu w lizatach komórek HeLa, z których 30 to dobrze znane białka wiążące alfa-ketoglutaran, co świadczy o wysokiej czułości i niezawodności metody. Ponadto w ramach projektu PELSA zidentyfikowano białka wiążące inne metabolity, takie jak kwas foliowy, leucyna, fumaran i bursztynian, co ukazuje jego szerokie zastosowanie.
PELSA może bezpośrednio wykryć wywołane ligandami zmiany stabilności lokalnej białek w całym lizacie komórkowym bez konieczności chemicznej modyfikacji ligandów. Ma szerokie zastosowanie do różnych ligandów i pozwala na systematyczną analizę białek wiążących ligandy, ich miejsc wiązania i lokalnego powinowactwa wiązania w lizacie komórkowym, gdzie białka niosą fizjologiczne modyfikacje potranslacyjne i są powiązane z oddziałującymi białkami.
Odniesienie: „Test stabilności lokalnej skoncentrowany na peptydach umożliwia identyfikację celów białkowych i regionów wiązania różnych ligandów w skali proteomu” autorstwa Kejia Li, Shijie Chen, Keyun Wang, Yan Wang, Lianji Xue, Yuying Ye, Zheng Fang, Jiawen Lyu , Haiyang Zhu, Yanan Li, Ting Yu, Feng Yang, Xiaolei Zhang, Siqi Guo, Chengfei Ruan, Jiahua Zhou, Qi Wang, Mingming Dong, Cheng Luo i Mingliang Ye, 10 grudnia 2024 r., Metody natury.
DOI: 10.1038/s41592-024-02553-7