Naukowcy z Scripps Research opracowali metodę wykorzystującą kodony czteronukleotydowe do włączania kodonów niekanonicznych aminokwasy w białka, poszerzając możliwości inżynierii białek bez konieczności edycji całego genomu. Metodę tę przetestowano pod kątem tworzenia nowych peptydów i ma ona potencjał do zastosowań w różnych dziedzinach.
Na każdych zajęciach wprowadzających z biologii jest to podstawowa koncepcja: białka składają się z kombinacji 20 różnych aminokwasów, ułożonych w różne sekwencje, jak słowa. Jednakże badacze chcący zaprojektować cząsteczki biologiczne o nowych funkcjach od dawna uznali, że te 20 elementów jest restrykcyjne. W rezultacie poszukiwali sposobów włączania do białek nowych składników – znanych jako aminokwasy niekanoniczne.
Teraz naukowcy z Scripps Research opracowali nowy paradygmat łatwego dodawania niekanonicznych aminokwasów do białek. Ich podejście, opisane w Biotechnologia Przyrody 11 września 2024 r. kręci się wokół używania czterech RNA nukleotydy – zamiast typowych trzech – do kodowania każdego nowego aminokwasu kwas.
Nowe podejście do inżynierii białek
„Naszym celem jest opracowanie białek o dostosowanych funkcjach do zastosowań w dziedzinach, od bioinżynierii po odkrywanie leków” – mówi starszy autor, dr Ahmed Badran, adiunkt chemii w Scripps Research. „Możliwość włączania niekanonicznych aminokwasów do białek za pomocą tej nowej metody przybliża nas do tego celu”.
Aby komórka mogła wyprodukować dowolne białko, musi przełożyć nić RNA na ciąg aminokwasów. Każde trzy nukleotydy RNA, zwane kodonami, odpowiadają jednemu aminokwasowi. Jednak wiele aminokwasów ma więcej niż jeden możliwy kodon; na przykład RNA odczytujący sekwencje UAU i UAC odpowiadają aminokwasowi tyrozynie. Zadaniem małych cząsteczek zwanych transferowymi RNA (tRNA) jest łączenie każdego aminokwasu z odpowiadającymi mu kodonami.
Niedawno badacze chcący dodać do białka zupełnie nowe aminokwasy opracowali strategie ponownego przypisania kodonu. Na przykład kodon UAU można połączyć z nowym aminokwasem poprzez zmianę tRNA na UAU; spowodowałoby to, że komórka odczytałaby UAU jako odpowiadający elementowi budulcowemu innemu niż tyrozyna. Ale jednocześnie każda instancja UAU w genomie komórki musiałaby stać się UAC, aby zapobiec integracji nowego aminokwasu z tysiącami innych białek, do których nie należy.
„Tworzenie wolnych kodonów poprzez przekodowanie całego genomu może być potężną strategią, ale może być również przedsięwzięciem wymagającym, ponieważ wymaga znacznych zasobów do zbudowania nowych genomów” – mówi Badran. „W przypadku samego organizmu przewidzenie, jak takie zmiany w kodonach wpływają na stabilność genomu i produkcję białek gospodarza, może być trudne”.
Wprowadzenie kodonów czteronukleotydowych
Badran i jego współpracownicy chcieli stworzyć skuteczną strategię typu plug-and-play, która obejmowałaby jedynie wybrane niekanoniczne aminokwasy w określonych miejscach docelowego białka, bez zakłócania normalnej biologii komórki lub wymagania całego genomu. edytowane. Oznaczało to użycie tRNA, które nie zostało jeszcze przypisane do aminokwasu. Ich rozwiązanie: czteronukleotydowy kodon.
Zespół wiedział, że w kilku sytuacjach – np. gdy bakterie szybko przystosowują się do oporności na leki – kodony czteronukleotydowe wyewoluowały w sposób naturalny. Dlatego w swojej nowej pracy naukowcy zbadali, co powoduje, że komórki używają kodonu zawierającego cztery nukleotydy zamiast trzech. Odkryli, że tożsamość sekwencji w pobliżu czterozasadowego kodonu ma kluczowe znaczenie — często używane kodony poprawiają zdolność komórki do odczytania czteronukleotydowego kodonu w celu włączenia niekanonicznego aminokwasu.
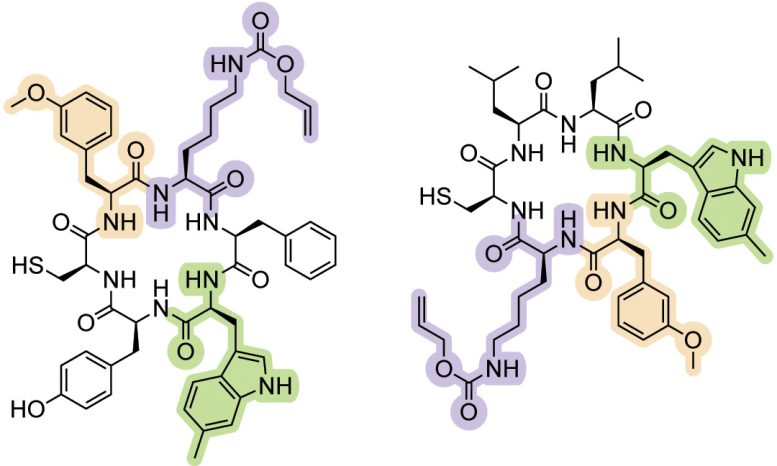
Następnie grupa Badrana przetestowała, czy można zmienić sekwencję pojedynczego genu, tak aby miał on nowy czteronukleotydowy kodon, który byłby prawidłowo wykorzystywany przez komórkę. Metoda zadziałała: kiedy badacze otoczyli miejsce docelowe trzyliterowymi, często używanymi kodonami i utrzymali wystarczający poziom czteronukleotydowego tRNA, komórka włączała każdy nowy aminokwas, który był przyłączony do odpowiedniego czteroliterowego tRNA. Zespół badawczy powtórzył eksperyment z 12 różnymi czteronukleotydowymi kodonami, a następnie wykorzystał tę technikę do zaprojektowania ponad 100 nowych cyklicznych peptydów – zwanych makrocyklami – zawierających maksymalnie trzy niekanoniczne aminokwasy w każdym.
„Te cykliczne peptydy przypominają bioaktywne małe cząsteczki, które można znaleźć w naturze” – mówi Badran. „Wykorzystując programowalność syntezy białek i różnorodność elementów składowych dostępnych dzięki temu podejściu, możemy stworzyć nowe w naturze małe cząsteczki, które będą miały ekscytujące zastosowania w odkrywaniu leków”.
Korzyści z nowej metody
Dodaje, że w porównaniu z poprzednimi podejściami do włączania niekanonicznych aminokwasów, ta nowa metoda jest łatwa w użyciu, ponieważ polega na zmianie tylko jednego genu, a nie całego genomu komórki. Ponadto w pojedynczym białku można zastosować więcej aminokwasów niekanonicznych, ponieważ istnieje więcej możliwych kodonów czteronukleotydowych niż trzynukleotydowych.
„Nasze wyniki sugerują, że obecnie można łatwo i skutecznie włączać niekanoniczne aminokwasy w różnych miejscach do szerokiej gamy białek” – mówi Badran. „Jesteśmy podekscytowani możliwościami związanymi z naszą bieżącą pracą i możliwością zapewnienia tej możliwości szerszej społeczności”.
Zauważa, że technikę tę można zastosować do przeprojektowania istniejących białek lub stworzenia zupełnie nowych białek, które będą przydatne w wielu sektorach, w tym w medycynie, przemyśle wytwórczym i wykrywaniu substancji chemicznych.
Odniesienie: „Efektywna ekspansja kodu genetycznego bez modyfikacji genomu gospodarza” autorstwa Alana Costello, Alexandra A. Petersona, Davida L. Lanstera, Zhiyi Li, Gavrieli D. Carvera i Ahmeda H. Badrana, 11 września 2024 r., Biotechnologia Przyrody.
DOI: 10.1038/s41587-024-02385-y
Zadanie to zostało wsparte środkami finansowymi ze środków m.in Narodowe Instytuty Zdrowia (DP5-OD024590), Research Corporation for Science Advancement, Sloan Foundation (G-2023-19625), Thomas Daniel Innovation Fund (627163_1), nagroda Abdul Latif Jameel Water and Food Systems Lab Grand Challenge Award (GR000141-S6241) , przełomowy grant Energy Explorer (GR000056), Foundation for Food & Agriculture Research (28-000578), Homeworld Collective Garden Grant (GR000129), Army Research Office (81341-BB-ECP), Hope Funds for Cancer Research (HFCR-23-03-01), stypendium Skaggs-Oxford i stypendium Fundacji Fletchera Jonesa.